近日,山东大学微生物技术国家重点实验室张伟教授与上海交通大学林厚文教授合作,在化学领域权威期刊《德国应用化学》(Angewandte Chemie International Edition, IF: 16.823)上发表题为“Somalactams A–D: Anti-inflammatory Macrolide Lactams with Unique Ring Systems from an Arctic Actinomycete Strain”的论文。张伟教授和上海交通大学林厚文教授为论文并列通讯作者,杨帆副研究员和微生物技术国家重点实验室桑茉莉博士生为论文共同第一作者。山东大学微生物技术国家重点实验室为第二完成单位和通讯作者单位。

深海来源的骨架新颖、机制独特天然产物的发现,对海洋药物的研究具有重要意义,是开发和利用深海生物资源的重要突破方向。作者以P450酶为探针,从北极海绵(水深200米)共附生放线菌Streptomyces somaliensis1107菌株基因组中挖掘获得含有2个P450酶编码基因的新颖基因簇sml,其中smlB-D编码聚酮合酶(PKS),smlA编码聚酮合酶-非核糖体多肽合成酶(PKS-NRPS)杂合蛋白。除此之外,该基因簇包含4个氧化还原酶编码基因,7个与前体单元合成酶编码基因,2个糖基转移酶编码基因,2个水解酶编码基因及负责转录调控和编码未知功能的基因。
通过系统的化学成分挖掘,从该菌株发酵液中分离得到4个极其罕见的21元新大环内酯类化合物Somalactams A-D(1-4),其中3个具有独特的多环稠合系统。综合运用高阶二维核磁、J B C A(基于耦合常数的构象分析),单晶衍射和化学修饰(Mosher)等方法确定了其平面与立体构型(图1)。生物活性研究表明化合物1在体外对LPS刺激的巨噬细胞RAW264.7释放的炎症因子IL-6和TNF抑制率IC50值为5.76 µM和0.18 µM并且无细胞毒性;药理研究表明化合物1通过抑制炎症相关通路MAPK调节下游蛋白的表达从而发挥抗炎作用且呈浓度依赖性。
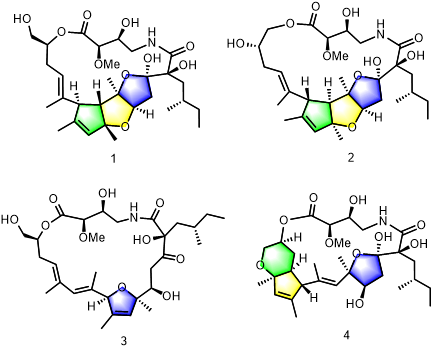
图1. SomalactamsA-D (1 - 4)化学结构
利用Crisper-Cas9介导的无痕基因敲除实验证实,sml基因簇负责somalactams类化合物的生物合成,P450酶Sml5参与了大环内酯骨架C-5的羟基化修饰。Somalactams的合成是由smlA-D的8个PKS模块和嵌入smlA中的1个NRPS模块形成的复合酶共同催化合成,其中,NRPS合成模块的底物预测为甘氨酸。基于somalactams的化学结构和对PKS底物识别功能域的氨基酸序列进行比对,该类化合物结构中的三个非常规结构单元分别由AT1、AT8和AT9负责识别和激活。其中,AT1识别和激活羟基乙酰辅酶A(glycolyl-CoA),AT8负责识别罕见的4-甲基己酰辅酶A(4-methylhexanoyl-CoA),AT9识别甲氧基丙二酸(methoxymalonyl-CoA)。聚酮链合成过程中,酮基还原酶(Ketoreductase,KR)、脱水酶(Dehydratase,DH)和烯基还原酶(enoyl reductase,ER)的数量和功能与somalactams化学结构之间存在严格的线性对应关系。基于以上分析,somalactams生物合成以非典型的glycolyl-CoA为起始单元,经过九步连续的脱羧缩合反应合成聚酮前体。根据somalactams的化学结构,推测化合物1 - 4是由SmlA末端的硫酯酶结构域(TE)催化聚酮链上的羟酰基单元发生α-OH或β-OH分子内环化,随后经P450酶Sml5等氧化酶的氧化、亲核环化及烯烃-环氧化等后修饰反应形成终产物(图2)。

图2. S. somaliensis 1107中sml生物合成基因簇及somalactams生物合成途径推
(①,α-OH参与的分子内环化;②,β-OH参与的分子内环化;a,环氧化物亲核环化;b,烯烃-环氧化物环化。中括号内为推测的合成中间体)。
该研究拓展了大环内酯类天然产物的结构骨架类型,提出了新的多环稠合的大环内酯类化合物生物合成机制,突显了深海微生物在发现结构新颖、活性独特的天然产物方面的重要价值,为创新深海药物或先导化合物的研究提供了新的范例。
本研究得到了国家重点项目研发计划、国家自然科学基金、山东省自然科学基金等科研项目的支持。
原文链接:https://onlinelibrary.wiley.com/doi/10.1002/anie.202218085

